2.黑龙江省农业科学院农药应用研究中心, 哈尔滨, 150086
 作者
作者  通讯作者
通讯作者
《分子植物育种》网络版, 2011 年, 第 9 卷, 第 77 篇 doi: 10.5376/mpb.cn.2011.09.0077
收稿日期: 2011年05月10日 接受日期: 2011年05月25日 发表日期: 2011年06月17日
李红艳等, 2011, 落叶松总DNA的提取及ISSR-PCR引物筛选, 分子植物育种 Vol.9 No.77 (doi: 10.5376/mpb.cn.2011.09.0077)
以针叶树落叶松当年生新梢为试材,对落叶松总DNA提取方法和ISSR-PCR引物筛选进行了试验。改良CTAB法可以在较短时间内提取到高纯度的DNA产物,用于ISSR-PCR扩增时其稳定性和重复性都很好。利用50个ISSR引物对来自黑龙江省林口县青山林场的10个个体进行PCR扩增,筛选出了多态性丰富、条带清晰且重复性良好的8个引物。本研究为落叶松种质资源的评估及遗传多样性分析奠定基础。
落叶松为松科落叶松属的落叶乔木,是我国东北、内蒙古林区以及华北、西南的高山针叶林的主要森林组成树种,是东北地区主要三大针叶用材林树种之一。ISSR (Inter-simple Sequence Repeats, 内部简单重复序列)分子标记技术是1994年Zietkiewicz等提出的,该技术具有模板DNA用量少,实验操作简单、快速、高效,遗传多态性高,重复性好等特点。已在多种动植物的种质鉴定(孙淑霞等, 2011;He et al., 2007)、遗传多样性分析(李志辉等, 2009; 杨传平等, 2005)、遗传作图和基因定位(马红勃等, 2008; 连莲, 2008)研究方面得到广泛应用。
高质量的总DNA及稳定的引物是获取可靠分子标记结果的前提,虽然已有对落叶松进行分子标记研究的若干报道(那冬晨等, 2006; 张磊等, 2008),但关于详细研究落叶松DNA提取方法的报道较少。因此,本文拟研究获得提取高质量落叶松总DNA的方法并筛选适合ISSR-PCR反应体系的引物,为进一步用ISSR技术进行落叶松生产中的品种快速鉴定和种质资源的保护利用奠定基础。
1结果与分析
1.1落叶松DNA的提取
改良CTAB法提取的落叶松基因组DNA主带清晰,无明显拖尾,未降解,无RNA,点样孔也没有过多的样品残留(图1),说明提取的DNA质量较高,完整性较好,没有较多的蛋白质等污染。紫外分光光度检测OD260/OD280值在1.75~1.95之间,表明DNA样品纯度较高,可用于ISSR引物的筛选分析和其他相关试验。
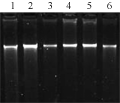
图1 部分样品基因组DNA电泳结果
注: 1~6号泳道分别为1~6号样品
Figure 1 Electrophoresis of genomic DNA
Note: Lane 1~6: Sample 1 to sample 6
1.2引物筛选
采用已知的落叶松ISSR-PCR的最适反应体系(林萍等, 2005),从50条引物中筛选出8条有效引物(表1),其扩增条带清晰、稳定、重复性好、多态性丰富。

表1 筛选出的8条ISSR引物
Table 1 Selected eight ISSR primers
1.3样品扩增
采用已知的落叶松ISSR-PCR的最适反应体系和筛选出的8条引物对林口青山的1~10号样品进行扩增,可见扩增条带清晰可辨,多态性丰富,差异明显(图2),说明本实验提取的DNA和引物筛选的质量较好,可以进行种质鉴定、遗传多样性分析等方面的研究。

图2 引物807, 809, 818, 849对1~10号样品扩增结果
Figure 2 Electrophoresis of 1~10 samples by primer 807, 809, 818, 849
2讨论
2.1 DNA提取方法改进
高纯度、高产率的基因组总DNA是分子生物学实验关键的第一步。不同的植物,提取的方法各异,对于同一植物,不同实验室所用仪器、试剂不同,提取方法也不尽相同。在实际应用中,要进行适当的改进方能获得较纯的产物。DNA的提取质量受许多因素影响,在提取基因组DNA过程中,各步骤微小的改变都能产生明显的效果。我们针对落叶松针叶不易磨碎、富含多酚、多糖、色素及蛋白质的特点,在CTAB法的基础上通过增加以下步骤提取总DNA。①在研磨时加入石英砂,可使液氮研磨针叶时能够尽可能地磨碎,保证其充分裂解;②向提取液中加入抗氧化剂β-巯基乙醇,可有效防止提取过程中材料与氧接触,自发氧化褐变或在多酚氧化酶(PPO)作用下发生酶促褐变,生成褐色的醌类物质等;③利用在乙醇的盐溶液中DNA的沉淀速度远远大于色素、多糖和酚类等其它物质的原理,在用无水乙醇沉淀DNA时加入低浓度乙酸钠,这样进一步去除了多糖、色素等其它杂质;④DNA沉淀后用RNase消化,排除了RNA的干扰。这种方法提取的DNA初提物呈白色,纯化后没有褐化现象,收率得以提高,经0.8%凝胶电泳和紫外分光光度计检测,DNA的质量和纯度都较好。
2.2 ISSR引物的筛选
试验以改进CTAB法提取的落叶松DNA为模板,对50条引物进行了筛选,共筛选出8条条带清晰、多态性丰富、重复性好的引物。通过引物的适合筛选,可以应用ISSR标记技术进行落叶松育种早期鉴定,为缩短育种年限、提高育种效率提供可能;可以快速、准确地分析落叶松不同群体之间以及同一群体内不同种质材料的遗传多样性水平和遗传分化程度,明确其亲缘关系,从而为落叶松种质资源鉴定、遗传图谱构建、分子标记辅助育种和基因工程等研究领域提供分子生物学依据。
3材料与方法
3.1材料
3.1.1试验材料
供试材料为落叶松当年生新梢,2009年6月采自黑龙江省林口县青山林场,置于冰上带回实验室,保存于超低温(-70℃)冰箱中。
3.1.2主要药品及试剂
(1)CTAB抽提缓冲液:2% CTAB(m/v),100 mmol/L Tris-HCl (pH 8.0),1.5 mol/L NaCl,20 mmol/L EDTA;(2)TE:10 mmol/L Tris-HCl;1.0 mmol/L EDTA;(3)氯仿:异戊醇=24:1;(4)β-巯基乙醇,TAE电泳缓冲液、琼脂糖、EB等。所用药品均为国产分析纯。(5)ISSR引物由上海生工(Sangon)合成,用于ISSR-PCR反应的Taq酶、dNTP以及DL 2000均为TaKaRa公司生产。
3.2方法
3.2.1 DNA的提取
用改进的CTAB法提取材料的总DNA。具体步骤如下:
①在1.5mL无菌离心管中加700 μL CTAB裂解液和20 μL的β-巯基乙醇,65℃浴热10 min。
②从-70℃冰箱中取出材料后用研钵加液氮和石英砂研磨成粉末状,向步骤①中的每个离心管中加约0.04 g,颠倒均匀,65℃恒温水浴30 min,其间定时温和摇匀。
③溶液冷至室温,加入700 μL的氯仿-异戊醇(24:1),轻轻振荡10 min。
④12 000 r/min离心10 min,取上清,加入氯仿-异戊醇,重复抽提2次。
⑤将上清中加入3滴3 mol/L醋酸钠和700 μL无水乙醇,沉淀30 min,12000 r/min离心15 min,弃上清。
⑥在沉淀中加入75%的乙醇700 μL清洗沉淀2次,12 000 r/min离心30 s后轻轻倒去乙醇。
⑦室温自然干燥离心管内的DNA沉淀,加入50 μL TE,使DNA充分溶解。
⑧加入2~3 μL RNase (10μg/μL),5 μL 10×Buffer,37℃下1~2 h。
⑨加入等体积氯仿,12 000 rpm离心5 min,取上清,加入3倍体积无水乙醇,混匀,12 000 rpm离心15 min,弃上清。
⑩室温气干,加入30 uL灭菌的去离子水,使DNA充分溶解。
3.2.2 DNA质量、纯度检测
取2 μL模板DNA在0.8%的琼脂糖凝胶中,于5 V/cm电压下电泳,检测总DNA的质量。利用Eppendorf紫外分光光度计(德国)测定DNA纯度和含量,以OD260值计算总DNA浓度,以OD260/OD280值判断总DNA的纯度。
3.2.3引物筛选方法
参照哥伦比亚大学公布的ISSR引物序列合成一系列引物,用林萍优化的落叶松ISSR-PCR反应体系(林萍等, 2005)分别进行扩增,从中筛选合适的引物。供试的ISSR引物序列情况见(表2)。

表2 试验所用引物及序列
Table 2 Primers and sequence
作者贡献
李红艳、罗旭及王丙锋是本研究的实验设计和实验研究的执行人;李红艳和王艳敏完成数据分析,论文初稿的写作;孙海滨参与实验设计,试验结果分析;李红艳是项目的构思者及负责人,指导实验设计,数据分析,论文写作与修改。全体作者都阅读并同意最终的文本。
致谢
本研究由黑龙江省自然科学基金项目(C200530)资助。
参考文献
He X.H., Li Y.R., Guo Y.Z., Ou S.J., and Li R.B., 2007, Identification of closely related mango cultivars by ISSR, Guihaia J., 27(1): 44-47
Li Z.H., Chen Y., Zhang D.L., Yang M.H., Jiang Y., Ding G.J., and Tan X.F., 2009, ISSR analysis of genetic diversity of pinnus massoniana on gupeng and langshui nature populations in guangxi, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 25(16): 116-119(李志辉, 陈艺, 张冬林, 杨模华, 蒋燚, 丁贵杰, 谭晓风, 2009, 广西马尾松天然林古蓬和浪水种源群体遗传多样性ISSR分析, 中国农学通报, 25(16): 116-119)
Lian L., 2008, Betula platyphylla linkage map ISSR AFLP pseudo-testcross, Thesis for M.S., Northeast Forestry University, Supervisor: Yan X.F., and Wei Z.G., pp.32-35 (连莲, 2008, 白桦ISSR和AFLP遗传图谱构建, 硕士学位论文, 东北林业大学, 导师: 阎秀峰, 魏志刚, pp.32-35)
Lin P., Zhang H.G., and Xie Y.H., 2005, Study on optimization for ISSR reaction system of larix using orthogonal design, Sengwu Jishu (Biotechnology), 15(5): 34-37 (林萍, 张含国, 谢运海, 2005, 正交设计优化落叶松ISSR-PCR反应体系, 生物技术, 15(5): 34-37)
Ma H.B., Qi J.M., Li Y.K., Liang J.X., Wang T., Lan T., Chen S., Tao A.F., Lin L.H., and Wu J.M., 2008, Construction of a molecular genetic linkage map of tobacco based on SRAP and ISSR markers, Zuowu Xuebao (Acta. Agronomica Sinica), 34(11): 1958-1963 (马红勃, 祁建民, 李延坤, 梁景霞, 王涛, 兰涛, 陈顺, 陶爱芬, 林荔辉, 吴建梅, 2008, 烟草SRAP和ISSR分子遗传连锁图谱构建, 作物学报, 34(11): 1958-1963)
Na D.C., Yang C.P., Jiang J., and Xia D.A., 2006, Analysis on the genetic diversity of larix gmelinii provenance by using ISSR markers, Linye Keji (Forestry Science & Technology), 31(1): 1-4 (那冬晨, 杨传平, 姜静, 夏德安, 2006, 利用ISSR标记分析兴安落叶松种源的遗传多样性, 林业科技, 31(1): 1-4)
Sun S.X., Li J., Chen D., Xie H.J., Tu M.Y., and Jiang G.L., 2011, Molecular identification of peach germplasm by ISSR Markers, Zhongguo Nongxue Tongbao (Chinese Agricultural Science Bulletin), 27(4): 173-177 (孙淑霞, 李靖, 陈栋, 谢红江, 涂美艳, 江国良, 2011, ISSR分子标记技术在桃品种鉴定中的应用, 中国农学通报, 27(4): 173-177)
Yang C.P., Wei L., Jiang J., Liu G.F., and Zhao G.Y., 2005, Analysis of genetic diversity for nineteen populations of pinus sibirica du tour with technique of ISSR, Dongbei Linye Daxue Xuebao (Journal of Northeast Forestry University), 33(1): 1-3 (杨传平, 魏利, 姜静, 刘桂丰, 赵光仪, 2005, 应用ISSR-PCR对西伯利亚红松19个种源的的遗传多样性分析, 东北林业大学学报, 33(1): l-3)
Zhang L., Zhang H.G., Li X.F., Jiang J., and Wei Z.G., 2008, Research on ISSR identification technology of larch species and clones, Zhiwu Yanjiu (Bulletin of Botanical Research), 28(2): 216-221 (张磊, 张含国, 李雪峰, 姜静, 魏志刚, 2008, 落叶松种间及无性系间ISSR鉴别技术的研究, 植物研究, 28(2): 216-221)
Zietkiewicz E., Rafalski A., and Labuda D., 1994, Genome fingerprinting by simple sequence repeat (SSR)-anchored polymerase chain reaction amplification, Genomics, 20(2):176-183
http://dx.doi.org/10.1006/geno.1994.1151 PMid:8020964